PARKIN-NANO
Nom du projet
Séquençage d’un biomarqueur de la maladie de PARKINson au travers d’un NANOcapteur
Acronyme du projet
PARKIN-NANO
Nom et prénom du porteur du projet
NICOLAÏ Adrien et DELARUE Patrice
Contacts
adrien.nicolai at u-bourgogne.fr
Etablissement
Université de Bourgogne
Laboratoire
Laboratoire Interdisciplinaire Carnot de Bourgogne (ICB)
Équipe de recherche
Département Nanosciences - Physique appliquée aux protéines
Résumé du projet
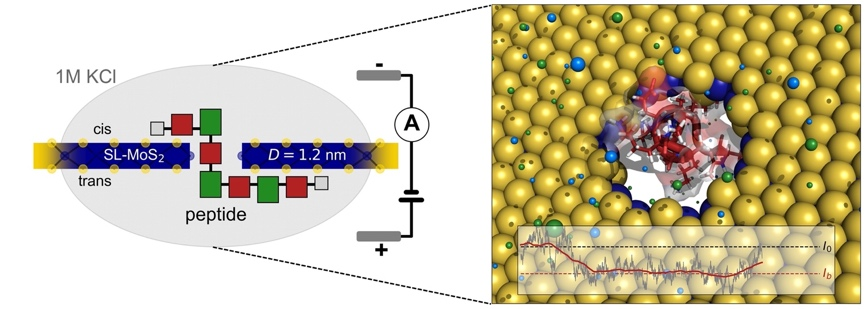
L’utilisation de capteur de type Nanopores 2-D solides pour la détection de la protéine alpha-Synucléine, biomarqueur de la maladie de Parkinson, présente un fort intérêt pour le diagnostic précoce de cette maladie. Dans ce projet, l’objectif était de modéliser à l’échelle atomique le nanocapteur complet et de simuler par dynamique moléculaire (DM) le passage complet de la protéine alpha-Synucléine (140 acides aminés) à travers le nanopore et d’enregistrer le signal de détection correspondant. Ces simulations ont permis de mettre en évidence comme preuve de concept l’utilisation de ces capteurs innovants de type MoS2 comme outil de détection précoce de la maladie de Parkinson.
Objectif initial du projet
L’objectif initial du projet, grâce aux moyens mis à disposition lors du MésoBFC – Défi CPU 2023 était la simulation de la détection complète du biomarqueur par le capteur. En effet, par le passé, seule la détection de « morceaux » de cette protéine alpha-Synucléine avait été réalisée (Nicolaï et al. Nanoscale 2020, 12, 22743-22753). De par la taille du système à modéliser puis simuler, le nombre de cœurs nécessaires est très important et les temps de simulations pour observer le passage complet de la protéine le sont également. Ce type de simulation de dynamique moléculaire « tous atomes » est donc un exploit en soit et n’a jamais été réalisée à notre connaissance sur une si longue protéine.
Pouvoir ainsi présenter plusieurs traces de courant ionique de la protéine alpha-Synucléine biomarqueur de la maladie de Parkinson lors de son passage dans le capteur nanopore de type MoS2 permet également de montrer à la communauté scientifique le type de trace attendue et ainsi de proposer une expérience en laboratoire qui pourrait permettre de confirmer ces observations.
Simulations et traitements réalisés
Les performances du logiciel GROMACS mesurés durant le défi sont d’environ 400 ns/jour sur 1392 cœurs. A titre de comparaison, les performances au Centre de calcul de l’uB sur des systèmes identiques et d’environ 50 ns/jour sur 256 cœurs.
Résultats obtenus
A partir des simulations de dynamique moléculaire réalisées dans ce projet, les traces de courant ionique enregistrées lors des 5 trajectoires (readout) par séquences ont été analysées. Ces traces sont disponibles en annexe (voir poster). A partir de ces traces de courant, l’objectif premier était de regarder la convergence pour déterminer si ces 5 traces étaient suffisamment cohérentes entre elles. Ce qui est le cas d’après notre étude. De plus, une transformation du signal de courant en signal « pas à pas » nous a permis d’identifier une quinzaine de niveau de courant caractéristiques (entre 10 et 17 selon les séquences) et plus d’une centaine d’exploration de ces niveaux par trajectoires. Sachant que la protéine alpha-Synucléine comprend 17 acides aminés différents dans sa composition pour un total de 140 acides aminés, nous pouvons à partir de ces résultats conclure que le capteur nanopore de type MoS2 est capable de séquencer la protéine (quasi) acide aminé par acide aminé avec une précision de lecture remarquable.
Une fois cette première étude menée, nous avons analysé en détail les signaux de courant et corréler les fluctuations observées avec la variation de volume de chaque acide aminé dans le nanopore. Cette seconde étude a permis de mettre en évidence que la mutation A30P n’influençait pas les propriétés du courant comparé à la protéine wild-type alors que la mutation E46K (mutation d’un acide aminé chargé négativement vers un acide aminé chargé positivement) avait un impact beaucoup plus important, que ce soit sur la durée de l’évènement de passage ou sa valeur relative de courant. D’autres variations le long de la séquence sont observées à différents endroits de la séquence, montrant que l’impact d’une mutation unique peut engendrée des modifications de courant à longue distance. Ces sites particuliers ont été identifiés et seront corrélés aux résultats structuraux obtenus dans l’équipe pour cette protéine dans une thèse précédente (Adrien Guzzo, 2022).
Enfin, les traces de courant le long de la séquence de la protéine alpha-Synucléine sont présentées en annexe (Poster, en bas de page) pour les trois séquences. Comme indiqué dans la section « objectif initial du projet », le type de trace attendue a bien été simulé et permet de proposer une expérience en laboratoire pouvant confirmer ces observations.
Valorisation des résultats
Communication des résultats : L’objectif à présent est la rédaction imminente d’un article scientifique sur les résultats obtenus pour les simulations réalisées lors du MésoBFC – Défi CPU 2023. De plus, les résultats seront communiqués pour la première fois sous format Poster lors de la conférence internationale S3IC 2024 - Single-Molecule Sensors and NanoSystems International Conference, qui se tiendra à Paris du 28 au 30 octobre 2024. Le poster qui sera présenté est disponible en annexe de ce rapport. Enfin, une communication orale des résultats est également envisagée par Adrien Nicolaï à la conférence internationale SMPS 2025 - Single Molecule Protein Sequencing, qui se tiendra à Bolzano, Italie du 18 au 24 janvier 2025.
Valorisation pédagogique : ces résultats seront présentés aux étudiants du Master international Physique, Photnoique et Nanotechnologies de l’université de Bourgogne. En effet, lors des travaux pratiques de NanoBioModelling, des simulations de dynamique moléculaire seront réalisés au CCuB sur des capteurs nanopores similaires au projet mais avec des petits morceaux de séquences.
Ouverture des donnés : Celle-ci est envisagée à la suite de la publication de l’article scientifique correspondant. Cela concernera principalement les données de courant ionique et trajectoires de la protéine à « gros grains ».